Soutenance de Thèse : Baptiste Alberti
| Quand ? |
Le 05/11/2025, de 14:00 à 17:00 |
|---|---|
| Où ? | Salle des thèses |
| S'adresser à | Baptiste Alberti |
| Participants |
Robin Andersson – Rapporteur – Associate Professor of Bioinformatics, Copenhagen University; Anamaria Necsulea – Rapportrice – Chargée de Recherche CNRS, LBBE; Olivier Gandrillon – Examinateur – Directeur de Recherche CNRS, LBMC; Arnau Sebe-Pedros – Examinateur – Group Leader, CRG, Barcelona; Marie Sémon – Examinatrice – Professeure à l’ENS de Lyon, LBMC; Yad Ghavi-Helm – Directrice de Thèse – Directrice de Recherche CNRS, IGFL; Paul Villoutreix – Co-Encadrant (membre du jury invité) – Chair de Professeur Junior INSERM, MMG, Marseille. |
| Ajouter un événement au calendrier |
|
Le 5 novembre, Baptiste Alberti de l'équipe de Yad Ghavi-Helm soutiendra sa thèse intitulée :
"Développer des outils bioinformatiques pour reconstruire l'activité spatio-temporelle tridimensionnelle des enhancers au cours de l'embryogenèse de Drosophila melanogaster"

Résumé :
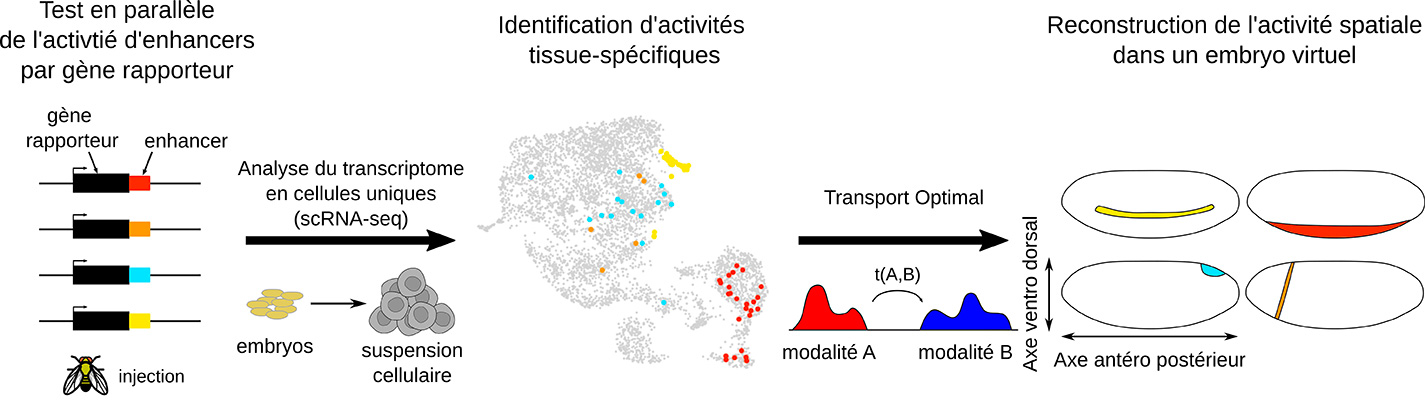
La régulation de l'expression des gènes au niveau transcriptionnel joue un rôle central dans la détermination du destin cellulaire et du développement en général. Les modules cis-régulateurs (CRMs) modulent cette expression en réponse à des signaux biologiques ou environnementaux. Ma thèse se concentre sur un type particulier de CRM : les enhancers, éléments d'ADN non codants régulant l'expression de leurs gènes cibles avec une grande précision spatiale et temporelle. Mon objectif était de développer une méthode pour cartographier cette activité à l’échelle de la cellule unique pour une multitude d’enhancers. En effet, bien que les avancées technologiques récentes aient amélioré la capacité à prédire la localisation des enhancers dans les génomes, leur activité spatiale et temporelle reste difficile à identifier car les tests d'activité des enhancers actuels souffrent soit d'un manque de débit, soit d'une résolution spatiale et temporelle insuffisante. Pour y remédier, nous avons développé spatial-scERA, une méthode combinant le séquençage d’ARN en cellule unique (scRNA-seq) avec des tests en parallèles d’activité d’enhancers et une reconstruction spatiale. Notre approche repose sur le transport optimal pour positionner les cellules des données scRNA-seq dans une reconstruction virtuelle de l'embryon en intégrant les profils transcriptomiques de ces cellules avec des connaissances a priori sur l'expression d'un ensemble de gènes marqueurs. Cela permet de cartographier l'activité des enhancers dans un tissu virtuel 3D reconstruit avec une résolution cellulaire. Nous avons utilisé spatial-scERA avec 25 enhancers candidats pour reconstruire leur activité dans un embryon virtuel de Drosophila au stade 6. Notre jeu de données inclut à la fois des enhancers déjà caractérisés et d’autres non annotés, permettant d’évaluer la robustesse de notre approche tout en identifiant de nouveaux éléments régulateurs. Comparée à des acquisitions d’HCR in situ, spatial-scERA reconstruit fidèlement l’activité de nos enhancers, y compris pour ceux détectés dans seulement 10 cellules dans les données scRNA-seq. Nous avons également montré que l'intégration des données spatiales permet de prédire les gènes cibles des enhancers et d’améliorer l'annotation des clusters scRNA-seq, permettant ainsi une identification plus précise des types cellulaires. Pour étendre notre méthode à d’autres stades de développement, nous avons généré et analysé des données scRNA-seq contenant les 25 mêmes enhancers aux stades 8 et 11. Nous avons d'abord étudié la dynamique d'activité des enhancers à partir des données scRNA-seq uniquement, en nous concentrant sur l'observation des variations de la spécificité tissulaire aux travers des 3 stades disponibles. Cependant, nous manquons actuellement d'un atlas 3D de l'expression des gènes marqueurs aux stades 8 et 11 pour produire une reconstruction. Pour générer cet atlas, nous développons la transcriptomique spatiale basée sur l'imagerie en utilisant la technologie MERFISH développé par Spatial-Cell-ID. Une fois que ces données seront disponibles, nous pourrons générer nos propres reconstructions à différents stades, et ainsi projeter nos enhancers des données scRNA-seq vers leurs positions spatiales dans un embryon. Notre approche offre un cadre puissant pour cartographier l’évolution spatio-temporelle de l’activité des enhancers. Applicable à tout organisme compatible avec des tests d’activité des enhancers et du scRNA-seq, notre méthode offre de nouvelles perspectives sur l’étude de la régulation des gènes dans les systèmes multicellulaires complexes.